https://github.com/DataSciBurgoon/ppar-gamma-model/blob/master/ppar-gamma_ligand_mlp.ipynb
DataSciBurgoon/ppar-gamma-model
PPAR-gamma deep learning model. Contribute to DataSciBurgoon/ppar-gamma-model development by creating an account on GitHub.
github.com
lyle박사님께서 구현해 놓은 PPAR-gamma 데이터를 이용하여 독성데이터 불균형 처리를 위한 방법에 대하여 알아보자.
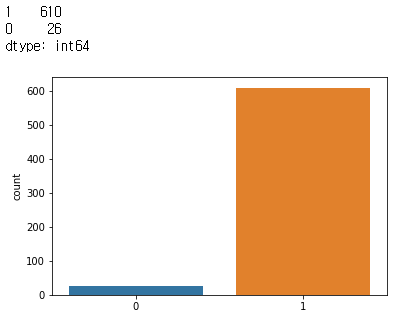
데이터분포

약4.2%의 ligand 95.8%의 not_ligand chemcial 존재. 굉장히 불균형(imbalanced)한 분포를 보인다.
Imbalanced Data를 처리하기 위한 방법
1. Use the right evaluation metrics
imbalanced data를 사용해서 model을 생성하게 되면 evaluation metrics를 부적절하게 해석 할 수 있기 때문에 위험하다. 지금 우리가 사용할 데이터의 경우도 not_lignad가 전체 데이터의 95.8%차지하기 때문에 전부 not_ligand로 판단한다고 해도 모델은 95.8%이상의 정확도를 보여줄 것이다. 하지만 이 정확도는 가치있는 정확도는 아니다.
이러한 경우에는 다른 대체 가능한 evalution metric를 사용하는 것이 좋다.
이 경우에는 recall즉 True Postive Rate 사용하는 것이 적절하다. TPR이 높아야 하는 것이다. 즉 실제 양성중 맞춘 양성의 비율이 높아야한다(실제 ligand중 맞춘 ligand의 비율이 높아야 한다)
ligand가 positive이고 not_ligand가 negative이다.
대체적으로 poistive가 적고 negative가 많다.
해깔릴때가 많은데
질병에 있어서 양성(볕 양)이라면 바이러스나 세균이 나타났다! 라고 생각하면 된다.
음성 이라면 바이러스가 침묵한다 즉 반응이 없다. 라고 생각하면 된다.
lyle박사님의 코드에 있는 데이터에 대해서 다시한번 정리하면
ligand는 positive이고 0으로 인코딩 되었다.
not_ligand는 negative이고 1로 인코딩 되었다.


sklearn.metrics의 confustion_matrix는 confusion_matrix(y_true, y_pred)과 같은 형태로 사용된다.

2. Resample the training set ( 훈련 데이터셋을 리샘플링한다)
undersampling과 over-sampling을 통해서 balanced dataset을 만들어 주는 방법이 있다.
2.1 Under-sampling
abundant class의 사이즈를 줄여서 balanced dataset으로 만드는 것이다. 이는 데이터의 양이 충분할때 사용할 수 있는 방법이다. rare class의 모든 샘플을 keeping하고 abundant class의 사이즈를 rare class와 같게 만든다. 랜덤으로 샘플을 선택해서 줄일 수도 있고 또 다른 여러가지 알고리즘 들이 있다.(https://datascienceschool.net/view-notebook/c1a8dad913f74811ae8eef5d3bedc0c3/)
2.2 Over-sampling
데이터의 양이 충분하지 않을때 사용한은 방법으로, rare sample의 사이즈를 증가시킨다. repetition, bootstrapping, SMOTE를 사용할 수 있다.
하나의 리샘플링 방법이 다른 리샘플링 방법보다 절대적인 이점은 없다. 이 두가지 방법의 적용은 데이터 세트에 따라 다르다. Over-, under-sampling을 조합하면 종종 좋은 결과를 보여준다.
3. Use K-fold Cross Validation in the right way ( k-flod cv를 옳은 방향으로 사용해야한다)
over-sampling을 사용할 때 적절한 방법. over-sampling은 rare samples의 distribution fuction을 기반으로 새로운 random data를 생성하기 위해서 bootstrapping을 적용하게된다. 만약 cross-validation이 over-sampling 이후에 적용이 되면, 기본적으로 하고자 하는것을 특정 artificial bootstrapping에 맞추는 것이다. cross-validation은 over-sampling을 하기 전에 항상 완료가 되어야 한다.
--> Lyle 박사님의 모델에 대해 내가 제시하고 싶은 문제점. 특정 artificial ligand chemical로 인해서 모델이 생성된다.
4. ensemble different resampled datasets (다른 재 샘플링된 데이터셋과 앙상블한다.)
더 많은 데이터를 사용해서 모델을 성공적으로 generalize하기 위한 쉬운 방법이다. 이 문제는 out-of-box classifier와 같은 logistic regression, random forest와 같이 rare class를 버리기 위해 generalize를 하는 경향이 있다. rare class의 모든 샘플들과, abundant class의 n-differing samples을 사용해서 n개의 models을 building 해 보자. 22개의 ensemble models이 만들어 질 것이다. (34개의 rare class, 762개의 abundant class), 762개를 22개의 chunks로 split을 해서 22개의 다른 모델을 train한다.
이 방법은 심플하고 데이터가 많아도 완벽하게 horizontally sclable(수평 확장성)하다. 또란 다른 클러스터의 노드들에서 각각의 모델을 학습할 수 있다는 것이 장점이다. ensemble models은 또한 generalize better, approach도 쉽다.
5. Resample with different ratios
이전의 approach는 rare와 abundant class 사이에 fine-tune이 가능하다. best ratio는 data에 의존적이고 그 모델은 사용이 된다. 그러나 ensemble에서 같은 ratio를 사용한 모든 모델을 train하는 대신에, different ratio를 ensemble하는 것이 더 좋다. 10개의 모델을 train했다면 하나는 1:1, 나머지는 1:3, 1:2로 사용된 모델에 따라 하나의 클래스가 얻는 가중치에 영향을 줄 수 있습니다.
6. abundant class를 r groups으로 clustering
training samples의 variety를 커버하기 위해서 random samples를 사용하는 대신에, clustering을 통해 abundant class를 r groups으로 clustering을 하는 것을 제안했다. r에 있는 classes의 개수를 r로, 각그룹에 대해서 medoid(centre of cluster)에 유지 된다. 그 모델은 rare class와 medoids로 부터 train 된다.
7. Design your own models
cost function을 디자인 해라.
만약 모델이 imbalanced data에 적잘하다면, resample은 필요가 없다. 유명한 XGBosst는 내부적으로 imbalanced가 되지 않도록 trains를 하기 때문에 이미 class가 너무 많이 skewed가 되지 않았다면 좋은 결과를 준다.
abundant class의 잘못된 classifcation보다 rare class의 잘못된 classification이 더 penalizing하다. cost function을 설계함으로써, rare class의 favour(편애)에서 naturally generalize를 할 수 있다
Lyle model의 경우
Lyle 박사님께서는 SMOTE를 사용하여 rare class의 sample size를 늘린 데이터셋을 input으로 사용했다. mlp모델엔 2개의 은닉층이 있고 classification을 위해 output layer의 activation function은 sigmoid를 사용했다. cost function은 binary_crossentropy를 사용했고 optimizer='adam'을 사용했다.
이러한 input_data와 model을 이용하여 5-fold cross validation을 했다.
논리적흐름에는 문제가 없지만. 내가 제시하고 싶은 문제점은 만들어진 최종 모델이 좋은 모델이 아니라는 것이다. 이는 새로운 데이터 예측에 매우 취약 할 수 있다. 근거를 제시 해 보면
- cross validation이 oversampling이후에 되었다.
train_set에만 oversampling이 적용 된 것이 아니라. 전체 데이터에 대해 oversampling 후 이를 train과 test로 나누었다.
이렇게 되면 기본적으로 하고자 하는 것이 샘플링된 artificial sample에 맞춰지는 방향이 된다. smote는 이미 존재하는 rare sample을 기반으로 이와 비슷한 artifical sample을 생성해 내는 과정이다. test_set에 있는 positive(0, ligand)물질은 train_set에 있는 물질과 매우 유사하여 test_set의 정보가 train_set으로 유출되었다고? 생각할수 있지 않을까?
따라서 새로운 사례의 데이터 예측에 매우 취약할 수 있다.
결론
lyle박사님의 논리 자체는 문제가 없다. 허나 이것이 좋은 모델은 아니다. 이는 새로운 데이터 예측에 매우 취약 할 수 있기 때문이다.
oversampling(smote) in all datasets
lyle 박사님이 제시한 모델 evaluation metrics은 TPR(Recall, Sensitivity) FPR(1-specificity)이다.
5-fold cv 결과는 다음과 같다.

| condition positive | condition negative | |
| predicted condition positive | TP(756) | FP(8) |
| predicted condition negative | FN(6) | TN(754) |
Recall = 756/762 --> ligand 예측성능
specificity = 754/762 --> not_ligand 예측성능
로 ligand의 예측성능, not_ligand의 예측성능 둘다 높다. 하지만 이 수치는 새로운 데이터를 예측시에 매우 달라진다
확인하기 위해 전체 데이터 셋에서 20%의 test data를 미리 떼어놓고 모델을 평가해 보자
no sampling

다음은 이 train_set그대로 모델을 학습하고 test_set으로 평가한 결과이다. lyle박사님께서 사용한 mlp모델을 그대로 가져왔다.
1. 0.95를 임계값으로 설정했을때
estimator = create_deep_learning_model()

2. kerasclassifier를 사용했을때결과estimator = KerasClassifier(build_fn=create_deep_learning_model,epochs=10, batch_size=10)

Recall = 0
specificity = 100
acc = 152/160 = 0.95
쓸모없는 모델이다. ligand인 물질을 전혀 찾아내지 못한다.
kerasclassifier에서 최적의 thershold 를 찾아주나?.. --> 확인해보기
oversampling(smote) only train dataset


다음은 train_set을 smote로 oversampling해서 모델을 학습하고 test_set으로 평가한 결과이다.

Randomly undersampling



데이터의 수가 굉장히 적으므로 5-fold cross validation 검증 하였다.
undersampling기법이 가장 좋은 성능을 보인다. recall이 70퍼센트에 육박한다.
+ class_weight 도 적용하면 더 좋은 성능 낼 수 있다.
kerasclassifier는 decision thershold를 0.5로 사용한다.
https://stackoverflow.com/questions/42606207/keras-custom-decision-threshold-for-precision-and-recall
Keras custom decision threshold for precision and recall
I'm doing a binary classification using Keras (with Tensorflow backend) and I've got about 76% precision and 70% recall. Now I want to try to play with decision threshold. As far as I know Keras uses
stackoverflow.com
다음 링크를 참조해서 코드를 구현하고 최적의 thershold를 찾아보자
design cost function
focal loss

https://github.com/umbertogriffo/focal-loss-keras
umbertogriffo/focal-loss-keras
Binary and Categorical Focal loss implementation in Keras. - umbertogriffo/focal-loss-keras
github.com
class weights

rare class에 높은 가중치,
abundant class에 낮은 가중치
이 weight가 뜻하는 것은 무엇인가?

가중치 (매개 변수) - 가중치는 단위 간의 연결 강도를 나타냅니다. 노드 1에서 노드 2까지의 가중치가 더 큰 경우, 뉴런 1이 뉴런 2보다 더 큰 영향을 미친다는 것을 의미합니다. 가중치는 입력 값의 중요성을 떨어 뜨립니다. 0에 가까운 가중치는 이 입력을 변경하면 출력을 변경하지 않음을 의미합니다. 음의 가중치는이 입력을 늘리면 출력이 감소 함을 의미합니다. 가중치는 입력이 출력에 미치는 영향을 결정합니다. -https://www.quora.com/What-does-weight-mean-in-terms-of-neural-networks
Weights(Parameters) — A weight represent the strength of the connection between units. If the weight from node 1 to node 2 has greater magnitude, it means that neuron 1 has greater influence over neuron 2. A weight brings down the importance of the input value. Weights near zero means changing this input will not change the output. Negative weights mean increasing this input will decrease the output. A weight decides how much influence the input will have on the output.
0(positive) 즉 ligand를 예측하기 위한 가중치에 더 큰 비중을 둔다. 0에 가가운 가중치는 입력이 변경되도 출력이 변경되지 않음을 의미한다.

Focal loss 를 사용해서 smote적용하지 않고 5-fold cross validation 한 결과

Recall = 0.0588, specificity = 0.9750, acc = 0.9359
-->별로 효과 없다.
focal loss 사용 & class weight 조정 해서 test 한 결과

-->효과 없다...
binary_crossentropy를 사용했을땐 굉장히 학습이 느리다.

1000epoch을 해도 under fitting이 된다. 무엇이 원일일까? -->너무 적은 rare class.. train_set에만 smote를 적용해보자
그런데 binary_focal_loss를 사용했을때는 학습이 상당히 빨라진다.

무엇이 원인일까?
smote_train_only & binary_crossentropy & class_weight


recall = 1
specificity = 0.6558
acc = 0.6687
epoch을 160으로 늘려보자 ㅎㅎ

180으로 늘려보자

200으로 늘려보자

220으로 늘려보자

perfect fiiting 될수록 recall은 감소하고 specificity는 늘어난다.
ligand의 개수가 너무 적어서 정확하다고 보기 힘들다 --> 5-fold로 검증해야할 필요성.
어쨌든 class_weight를 조정하는것은 recall을 올리는데 효과가 좋다.
bagging boosting 효과 없다..

